Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunología
PatologíaTecnologíaIndustria
Eventos

- Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
- Nuevo método de análisis de sangre detecta opioides potentes en menos de tres minutos
- Kit inalámbrico de prueba de hepatitis B completa detección y recopilación de datos en un solo paso
- Dispositivo sensible, indoloro, de bajo costo y sin radiación detecta cáncer de mama en orina
- Prueba de saliva detecta cáncer de mama en cinco segundos
- Análisis de sangre identifica a personas con mayor riesgo de morir por insuficiencia cardíaca
- Análisis de sangre muestra precisión del 83 % para detectar cáncer colorrectal
- Análisis de sangre podría predecir adultos jóvenes que podrían desarrollar enfermedades del envejecimiento
- Prueba de diagnóstico molecular predice con precisión resultados de terapia en pacientes con cáncer de mama
- Novedosa tecnología de biosensor POC de molécula pequeña podría revolucionar detección temprana de enfermedades
- Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
- Rastreador portátil de glóbulos blancos podría permitir pruebas rápidas de infecciones
- Analizador hematológico optofluídico inteligente del tamaño de la palma de la mano permite realizar pruebas POC de células sanguíneas del paciente
- Plataforma de hematología automatizada ofrece desempeño analítico de alto rendimiento
- Nueva herramienta analiza plaquetas sanguíneas de forma más rápida, sencilla y precisa
- Técnicas de vanguardia para investigar respuesta inmune en infecciones mortales por estreptococo A
- Novedosos inmunoensayos permiten diagnóstico temprano del síndrome antifosfolípido
- Nueva prueba podría predecir éxito de inmunoterapia para una gama más amplia de cánceres
- Pruebas simples de proteínas en sangre predicen resultados de CAR T para pacientes con linfoma
- Tecnología de chip clasificador de células podría allanar el camino para elaboración de perfiles inmunológicos POC
- Nuevo análisis de sangre reduce tiempo de diagnóstico de infecciones por micobacterias no tuberculosas de meses a horas
- Nuevo análisis para tuberculosis podría ampliar acceso a pruebas en países de ingresos bajos y medios
- Prueba rápida diagnostica enfermedades tropicales en horas para tratamiento con antibióticos más rápido
- Pruebas moleculares rápidas permiten tratamiento antibiótico más rápido y específico para neumonía
- Plataforma rápida de PSA proporciona resultados terapéuticos específicos días antes que el estándar de atención actual
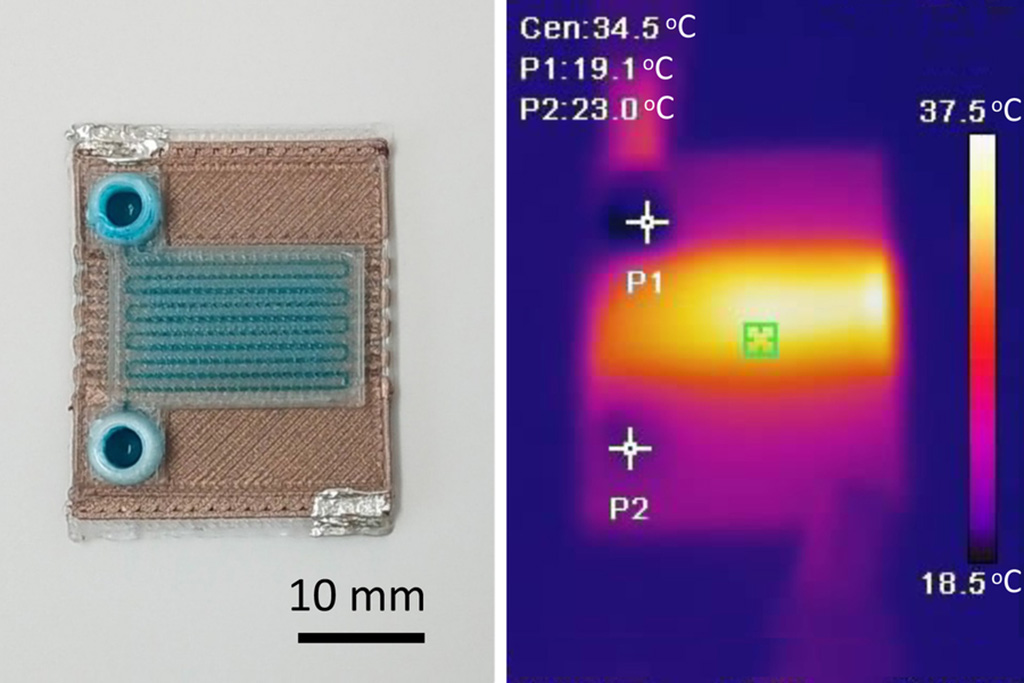
- Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
- Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
- Primera tecnología de su tipo para detectar glucosa en saliva humana
- Dispositivo electroquímico identifica personas con mayor riesgo de osteoporosis mediante una sola gota de sangre
- Nueva prueba no invasiva detecta infección por malaria sin muestra de sangre
- Beckman Coulter y Fujirebio amplían su asociación para diagnóstico de enfermedades neurodegenerativas
- Sysmex e Hitachi colaboran en desarrollo de nuevos sistemas de pruebas genéticas
- Sysmex y CellaVision amplían su colaboración para avanzar en soluciones de hematología
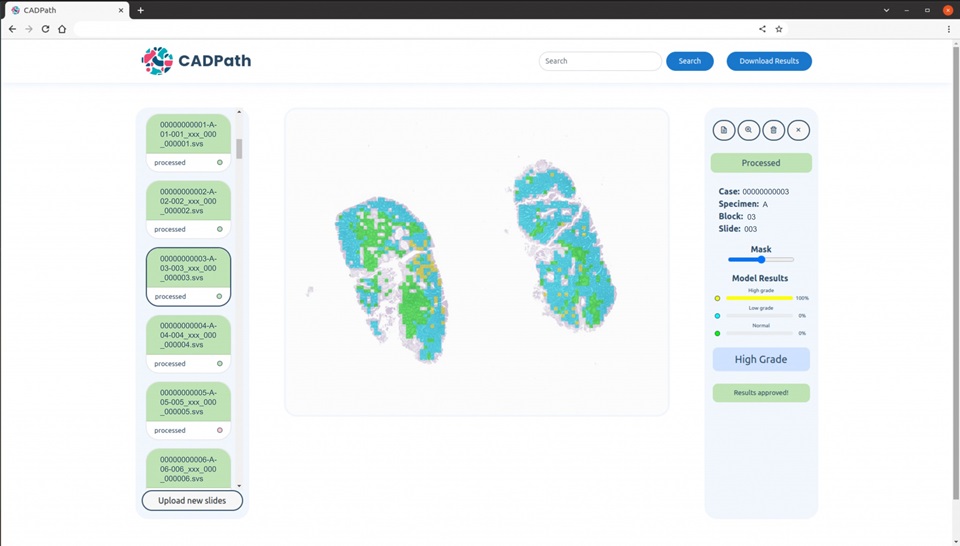
- BD y Techcyte colaboran en un sistema de citología cervical digital basado en IA para pruebas de Papanicolaou
- Medlab Middle East 2024 abordará potencial transformador de la inteligencia artificial
- IA supera a patólogos expertos en predicción de propagación del cáncer de pulmón
- Primer prototipo para aplicar IA al diagnóstico colorrectal
- Nueva prueba identifica cánceres de ovario agresivos más temprano
- Instrumento compacto de detección fotoacústica mejora diagnóstico biomédico de tejidos
- Dispositivo de esponja en cápsula no endoscópica ayuda a detectar cáncer de esófago

Expo
 ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunología
PatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunología
PatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros


- Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
- Nuevo método de análisis de sangre detecta opioides potentes en menos de tres minutos
- Kit inalámbrico de prueba de hepatitis B completa detección y recopilación de datos en un solo paso
- Dispositivo sensible, indoloro, de bajo costo y sin radiación detecta cáncer de mama en orina
- Prueba de saliva detecta cáncer de mama en cinco segundos
- Análisis de sangre identifica a personas con mayor riesgo de morir por insuficiencia cardíaca
- Análisis de sangre muestra precisión del 83 % para detectar cáncer colorrectal
- Análisis de sangre podría predecir adultos jóvenes que podrían desarrollar enfermedades del envejecimiento
- Prueba de diagnóstico molecular predice con precisión resultados de terapia en pacientes con cáncer de mama
- Novedosa tecnología de biosensor POC de molécula pequeña podría revolucionar detección temprana de enfermedades
- Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
- Rastreador portátil de glóbulos blancos podría permitir pruebas rápidas de infecciones
- Analizador hematológico optofluídico inteligente del tamaño de la palma de la mano permite realizar pruebas POC de células sanguíneas del paciente
- Plataforma de hematología automatizada ofrece desempeño analítico de alto rendimiento
- Nueva herramienta analiza plaquetas sanguíneas de forma más rápida, sencilla y precisa
- Técnicas de vanguardia para investigar respuesta inmune en infecciones mortales por estreptococo A
- Novedosos inmunoensayos permiten diagnóstico temprano del síndrome antifosfolípido
- Nueva prueba podría predecir éxito de inmunoterapia para una gama más amplia de cánceres
- Pruebas simples de proteínas en sangre predicen resultados de CAR T para pacientes con linfoma
- Tecnología de chip clasificador de células podría allanar el camino para elaboración de perfiles inmunológicos POC
- Nuevo análisis de sangre reduce tiempo de diagnóstico de infecciones por micobacterias no tuberculosas de meses a horas
- Nuevo análisis para tuberculosis podría ampliar acceso a pruebas en países de ingresos bajos y medios
- Prueba rápida diagnostica enfermedades tropicales en horas para tratamiento con antibióticos más rápido
- Pruebas moleculares rápidas permiten tratamiento antibiótico más rápido y específico para neumonía
- Plataforma rápida de PSA proporciona resultados terapéuticos específicos días antes que el estándar de atención actual
- Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
- Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
- Primera tecnología de su tipo para detectar glucosa en saliva humana
- Dispositivo electroquímico identifica personas con mayor riesgo de osteoporosis mediante una sola gota de sangre
- Nueva prueba no invasiva detecta infección por malaria sin muestra de sangre
- Beckman Coulter y Fujirebio amplían su asociación para diagnóstico de enfermedades neurodegenerativas
- Sysmex e Hitachi colaboran en desarrollo de nuevos sistemas de pruebas genéticas
- Sysmex y CellaVision amplían su colaboración para avanzar en soluciones de hematología
- BD y Techcyte colaboran en un sistema de citología cervical digital basado en IA para pruebas de Papanicolaou
- Medlab Middle East 2024 abordará potencial transformador de la inteligencia artificial
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- IA supera a patólogos expertos en predicción de propagación del cáncer de pulmón
- Primer prototipo para aplicar IA al diagnóstico colorrectal
- Nueva prueba identifica cánceres de ovario agresivos más temprano
- Instrumento compacto de detección fotoacústica mejora diagnóstico biomédico de tejidos
- Dispositivo de esponja en cápsula no endoscópica ayuda a detectar cáncer de esófago

















.jpg)

.jpg)
.jpg)



.jpg)

.jpg)