Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunologíaMicrobiologíaPatología
Industria
Eventos
Webinars

- Discrepancia entre dos pruebas comunes de función renal indica problemas de salud graves
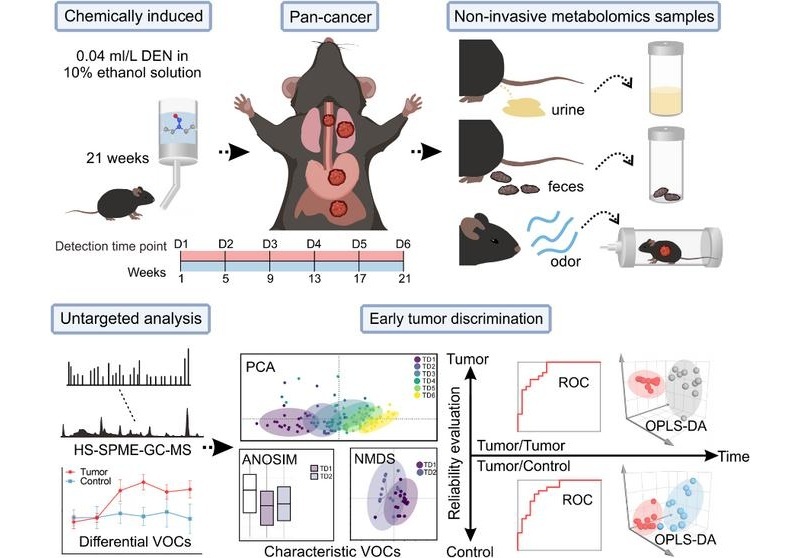
- COV son prometedores para detección temprana de múltiples cánceres
- Espectroscopia Raman portátil ofrece diagnóstico rentable de enfermedad renal en POC
- Nanopartículas de oro mejoran precisión del diagnóstico de cáncer ovárico
- Tecnología de aislamiento celular simultáneo mejora precisión del diagnóstico del cáncer
- Prueba sanguínea de cuatro genes descarta infección pulmonar bacteriana
- Nueva prueba PCR mejora precisión diagnóstica de vaginosis bacteriana y vaginitis por Candida
- Nueva estrategia de edición de marcadores séricos mejora diagnóstico de enfermedades neurológicas
- Primera prueba genética de riesgo de diabetes tipo 1 permite detección temprana
- Análisis sanguíneo evita cirugías innecesarias en pacientes con cáncer gástrico de bajo riesgo
- Medición de microvesículas podría detectar lesiones vasculares en pacientes con anemia falciforme
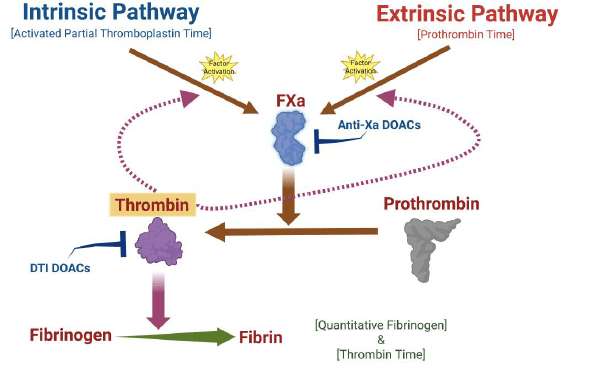
- Nueva guía de ADLM sobre pruebas de coagulación mejora atención a pacientes que toman anticoagulantes
- Pruebas viscoelásticas podrían mejorar tratamiento de hemorragia materna
- Modelo mide exposición a radiación en sangre para tratamientos precisos contra cáncer
- Las plaquetas podrían mejorar detección temprana y mínimamente invasiva del cáncer
- Prueba de firma genética predice respuesta a tratamientos clave para cáncer de mama
- Chip captura células cancerosas de sangre para seleccionar tratamiento adecuado para cáncer de mama
- Modelo de biopsia líquida en sangre analiza eficacia de inmunoterapia
- Genes característicos predicen expansión de células T en inmunoterapia
- Diagnóstico por microscopio molecular evalúa rechazo del trasplante de pulmón
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Sensor de saliva con IA permite detección precoz del cáncer de cabeza y cuello
- Tecnología de biosensores con IA detecta cáncer de pulmón mediante pruebas de aliento
- Modelo de IA detecta cáncer de ovario con precisión sin precedentes
- Biosensor portátil diagnostica trastornos psiquiátricos mediante muestras de saliva
- Dispositivo de clasificación celular utiliza levitación electromagnética para dirigir con precisión movimiento celular
- Roche y Freenome colaboran para desarrollar pruebas de detección del cáncer
- Co-Diagnostics crea nueva unidad de negocio para desarrollar diagnósticos con IA
- Qiagen adquiere empresa de ómica unicelular Parse Biosciences
- Puritan Medical Products presenta sus innovaciones en AMP2025 en Boston
- Advanced Instruments se fusionó como Nova Biomedical
- Herramienta de IA analiza rápidamente imágenes complejas de cáncer para tratamiento personalizado
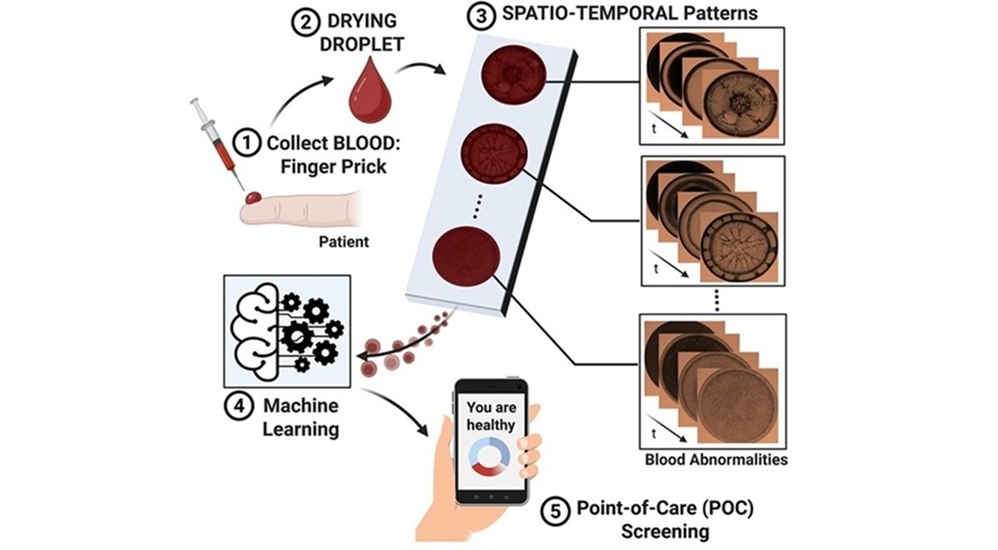
- Tecnología diagnóstica analiza rápidamente biofluidos utilizando una sola gota
- Nueva tecnología rastrea células cancerosas ocultas más rápido
- Herramienta de IA mejora detección del cáncer de mama
- Herramienta de IA predice éxito del tratamiento de cáncer de recto

 Expo
Expo
- Discrepancia entre dos pruebas comunes de función renal indica problemas de salud graves
- COV son prometedores para detección temprana de múltiples cánceres
- Espectroscopia Raman portátil ofrece diagnóstico rentable de enfermedad renal en POC
- Nanopartículas de oro mejoran precisión del diagnóstico de cáncer ovárico
- Tecnología de aislamiento celular simultáneo mejora precisión del diagnóstico del cáncer
- Prueba sanguínea de cuatro genes descarta infección pulmonar bacteriana
- Nueva prueba PCR mejora precisión diagnóstica de vaginosis bacteriana y vaginitis por Candida
- Nueva estrategia de edición de marcadores séricos mejora diagnóstico de enfermedades neurológicas
- Primera prueba genética de riesgo de diabetes tipo 1 permite detección temprana
- Análisis sanguíneo evita cirugías innecesarias en pacientes con cáncer gástrico de bajo riesgo
- Medición de microvesículas podría detectar lesiones vasculares en pacientes con anemia falciforme
- Nueva guía de ADLM sobre pruebas de coagulación mejora atención a pacientes que toman anticoagulantes
- Pruebas viscoelásticas podrían mejorar tratamiento de hemorragia materna
- Modelo mide exposición a radiación en sangre para tratamientos precisos contra cáncer
- Las plaquetas podrían mejorar detección temprana y mínimamente invasiva del cáncer
- Prueba de firma genética predice respuesta a tratamientos clave para cáncer de mama
- Chip captura células cancerosas de sangre para seleccionar tratamiento adecuado para cáncer de mama
- Modelo de biopsia líquida en sangre analiza eficacia de inmunoterapia
- Genes característicos predicen expansión de células T en inmunoterapia
- Diagnóstico por microscopio molecular evalúa rechazo del trasplante de pulmón
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Sensor de saliva con IA permite detección precoz del cáncer de cabeza y cuello
- Tecnología de biosensores con IA detecta cáncer de pulmón mediante pruebas de aliento
- Modelo de IA detecta cáncer de ovario con precisión sin precedentes
- Biosensor portátil diagnostica trastornos psiquiátricos mediante muestras de saliva
- Dispositivo de clasificación celular utiliza levitación electromagnética para dirigir con precisión movimiento celular
- Roche y Freenome colaboran para desarrollar pruebas de detección del cáncer
- Co-Diagnostics crea nueva unidad de negocio para desarrollar diagnósticos con IA
- Qiagen adquiere empresa de ómica unicelular Parse Biosciences
- Puritan Medical Products presenta sus innovaciones en AMP2025 en Boston
- Advanced Instruments se fusionó como Nova Biomedical
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Herramienta de IA analiza rápidamente imágenes complejas de cáncer para tratamiento personalizado
- Tecnología diagnóstica analiza rápidamente biofluidos utilizando una sola gota
- Nueva tecnología rastrea células cancerosas ocultas más rápido
- Herramienta de IA mejora detección del cáncer de mama
- Herramienta de IA predice éxito del tratamiento de cáncer de recto