Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Webinars

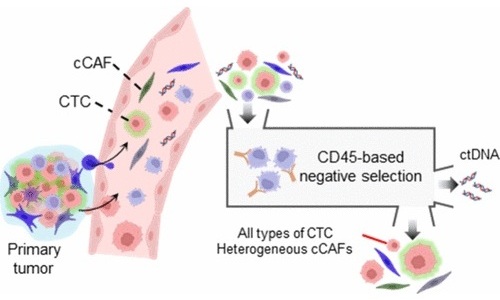
- Tecnología de aislamiento celular simultáneo mejora precisión del diagnóstico del cáncer
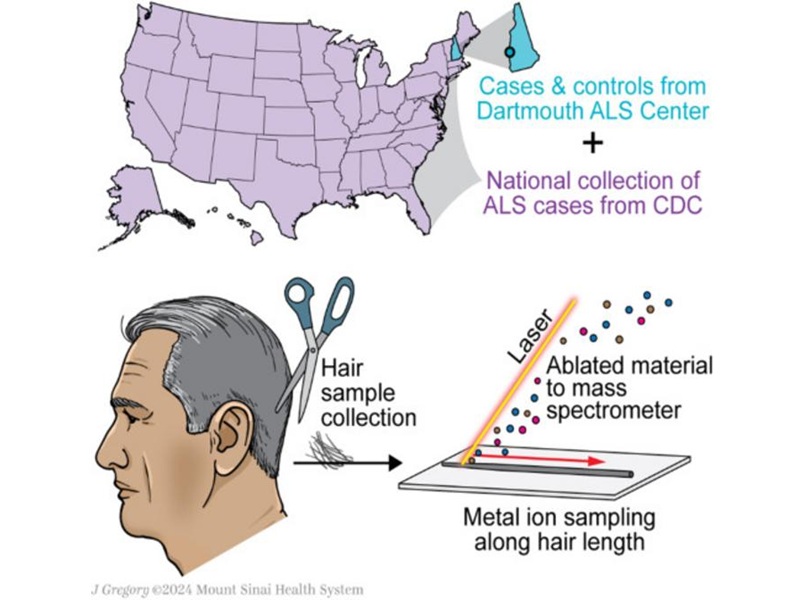
- Sencilla prueba capilar no invasiva podría acelerar diagnóstico de ELA
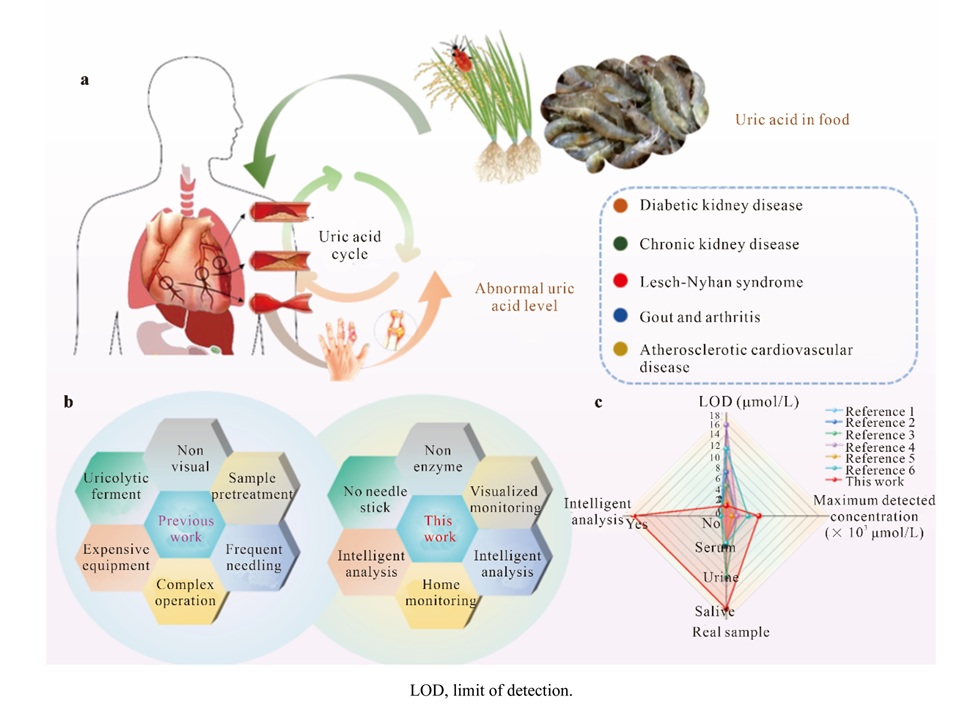
- Prueba de saliva detecta niveles elevados de ácido úrico sin necesidad de extraer sangre
- Marcadores de cáncer de próstata basados en composición química de calcificaciones aceleran detección
- Prueba de aliento ayuda a detectar cánceres de sangre
- Prueba genética predice respuesta a medicamentos para bajar de peso
- Prueba de tuberculosis con CRISPR utiliza hisopo bucal para simplificar detección
- Método basado en metilación del ADN predice progresión del cáncer
- Prueba de orina podría predecir resultado en trasplante de cartílago
- Análisis sanguíneo de cáncer en 2 horas transforma detección de tumores
- Modelo mide exposición a radiación en sangre para tratamientos precisos contra cáncer
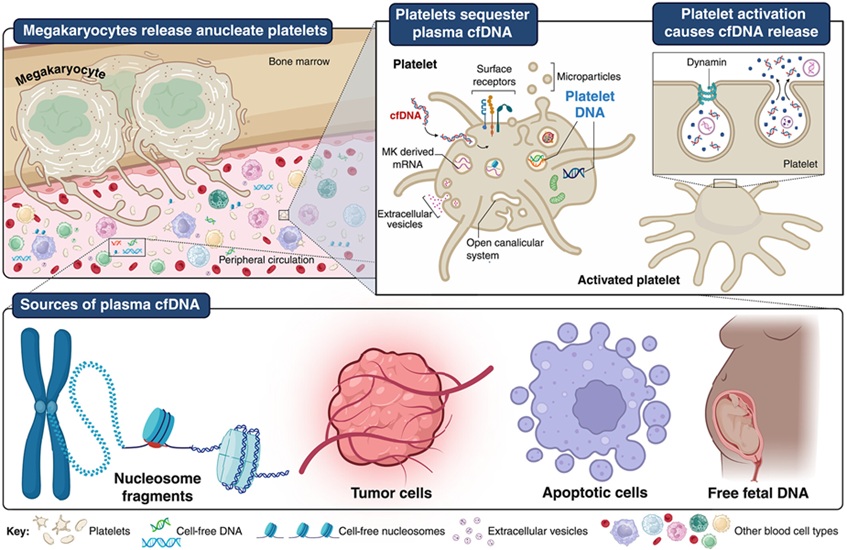
- Las plaquetas podrían mejorar detección temprana y mínimamente invasiva del cáncer
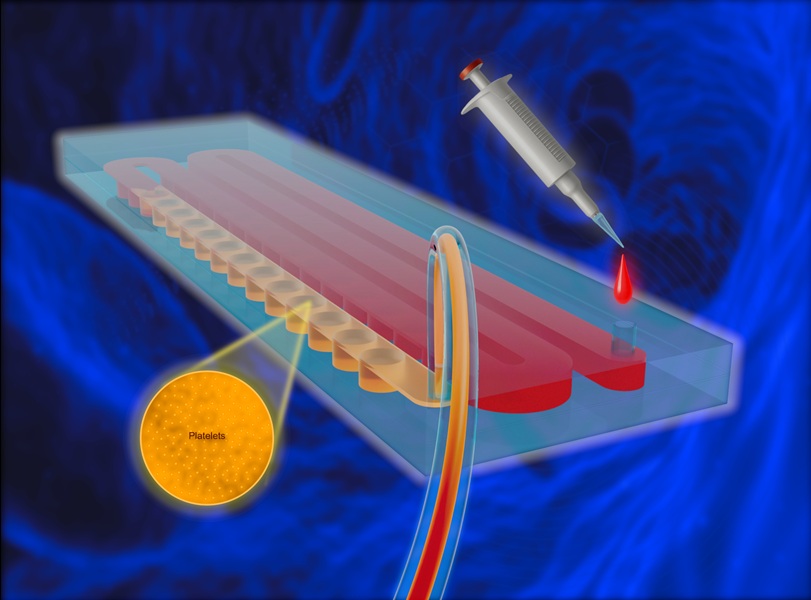
- Dispositivo portátil desechable obtiene plasma rico en plaquetas sin equipos complejos
- Prueba de cartucho desechable ofrece resultados de hemograma rápidos y precisos
- Primera prueba de monitorización de heparina POC proporciona resultados rápidos
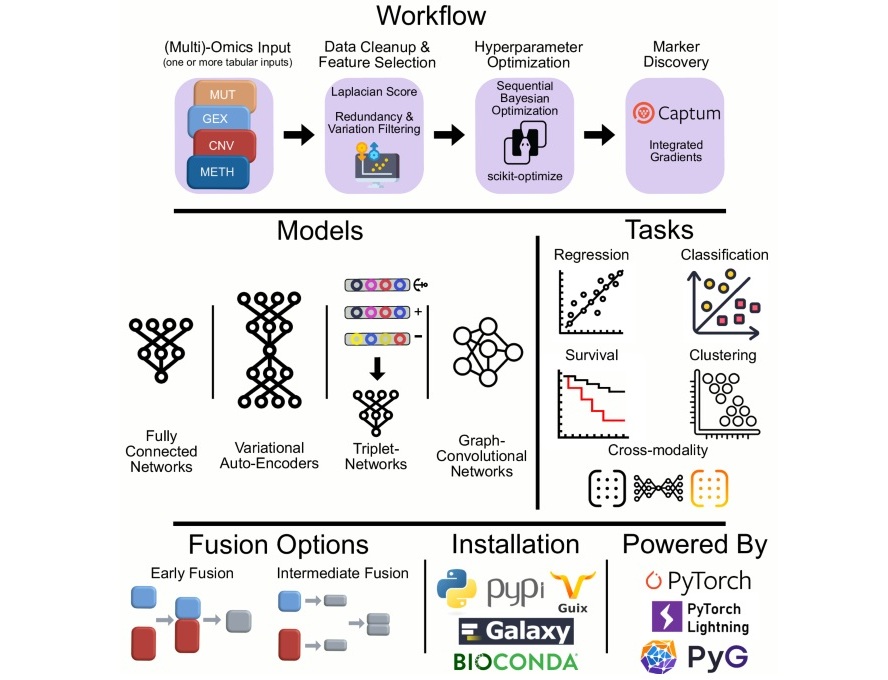
- Nueva herramienta utiliza aprendizaje profundo para terapia de precisión contra cáncer
- Prueba diagnóstica complementaria identifica cáncer de mama y de vías biliares con HER2 ultrabajo
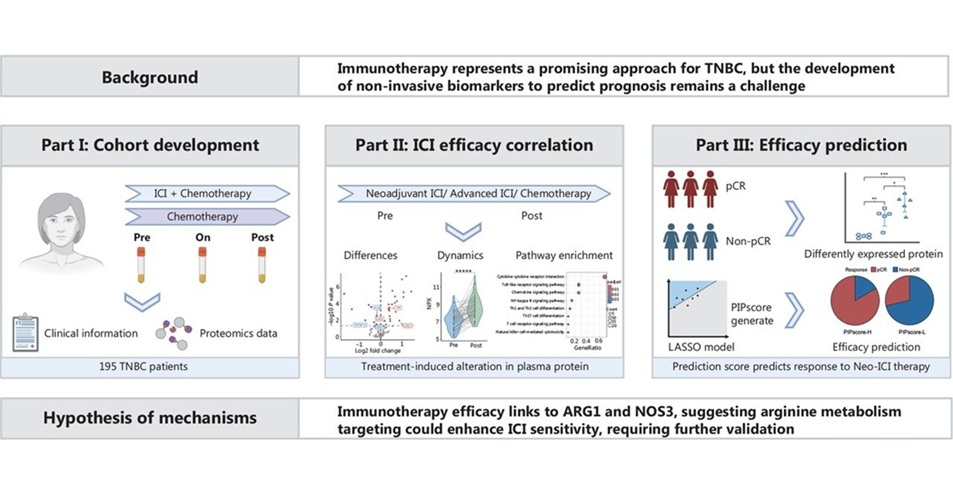
- Análisis sanguíneo predice eficacia de inmunoterapia en cáncer mamario triple negativo
- Pruebas genéticas simples podrían predecir éxito del tratamiento de esclerosis múltiple
- Nueva firma genética predice respuesta a inmunoterapia en cánceres renales avanzados
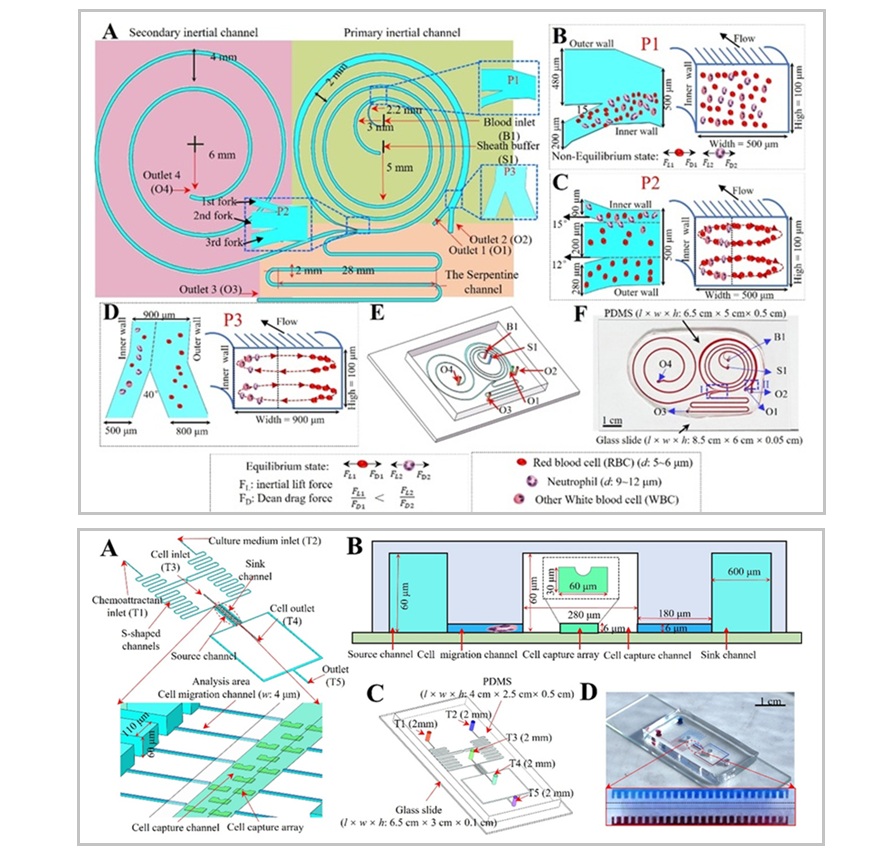
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
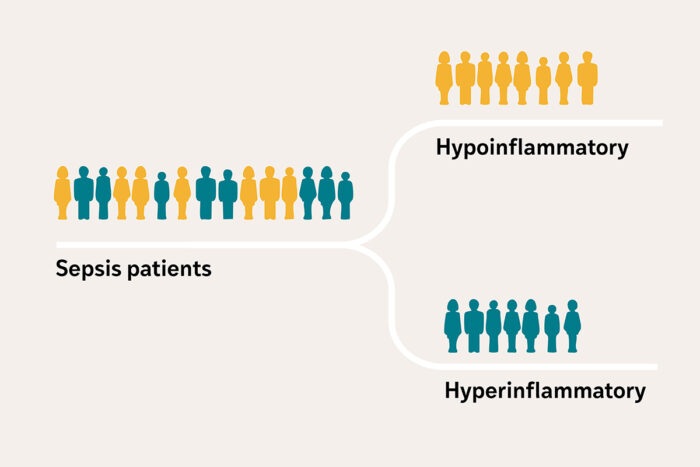
- Nuevo método diagnóstico confirma sepsis de forma más temprana
- Nuevos marcadores podrían predecir riesgo de infección grave por clamidia
- Espectroscopia portátil detecta de forma rápida y no invasiva bacterias en fluido vaginal
- Prueba de saliva basada en CRISPR detecta tuberculosis en esputo
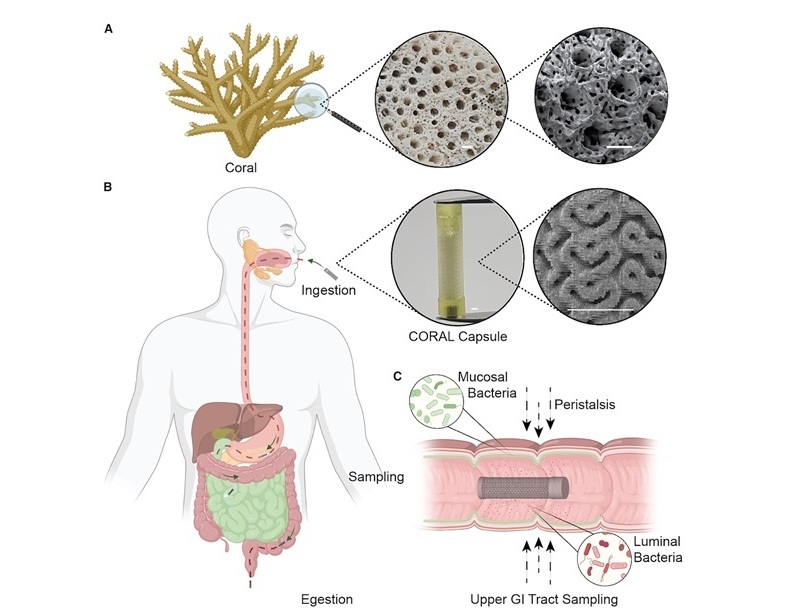
- Muestras de cápsulas inspiradas en corales ocultan bacterias intestinales
- Tecnología de diagnóstico rápido detecta infecciones del tracto respiratorio inferior en muestras de aliento
- Sensor de grafeno utiliza muestra de aliento para identificar diabetes y prediabetes en minutos
- Parche de sudor inalámbrico podría usarse como prueba diagnóstica para fibrosis quística
- Nuevo método mejora fiabilidad de la IA con aplicaciones en diagnóstico médico
- VedaBio se asocia con Mammoth Biosciences para expandir diagnósticos basados en CRISPR
- Werfen y VolitionRx se asocian para adelantar pruebas diagnósticas del síndrome antifosfolípido
- Siemens Healthineers y Carna Health se asocian para impulsar innovación en cuidado renal
- Tempus AI adquiere empresa de patología digital Paige
- Vircell lanza nuevo sitio web para sus productos de control molecular
- Adelanto en VSG amplía estabilidad de muestras sanguíneas de 4 a 28 horas
- Análisis patológico preciso mejora resultados del tratamiento del fibrosarcoma en adultos
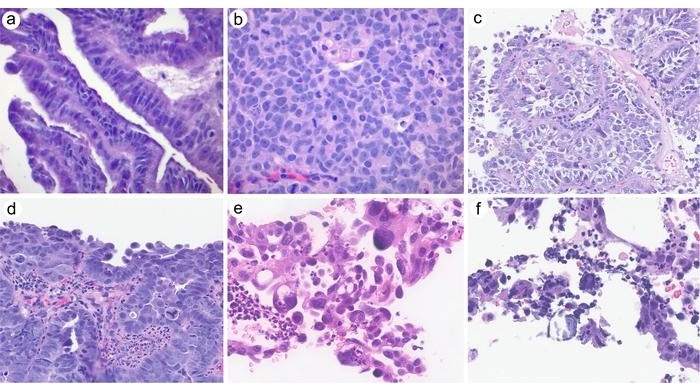
- Estudio clinicopatológico respalda exclusión del carcinoma seroso cervical de clasificación de OMS
- Sistema con IA compatible con dispositivos móviles revolucionará diagnóstico de malaria
- Microscopio compacto con IA permite puntuación del cáncer rápida y rentable

 Expo
Expo
- Tecnología de aislamiento celular simultáneo mejora precisión del diagnóstico del cáncer
- Sencilla prueba capilar no invasiva podría acelerar diagnóstico de ELA
- Prueba de saliva detecta niveles elevados de ácido úrico sin necesidad de extraer sangre
- Marcadores de cáncer de próstata basados en composición química de calcificaciones aceleran detección
- Prueba de aliento ayuda a detectar cánceres de sangre
- Prueba genética predice respuesta a medicamentos para bajar de peso
- Prueba de tuberculosis con CRISPR utiliza hisopo bucal para simplificar detección
- Método basado en metilación del ADN predice progresión del cáncer
- Prueba de orina podría predecir resultado en trasplante de cartílago
- Análisis sanguíneo de cáncer en 2 horas transforma detección de tumores
- Modelo mide exposición a radiación en sangre para tratamientos precisos contra cáncer
- Las plaquetas podrían mejorar detección temprana y mínimamente invasiva del cáncer
- Dispositivo portátil desechable obtiene plasma rico en plaquetas sin equipos complejos
- Prueba de cartucho desechable ofrece resultados de hemograma rápidos y precisos
- Primera prueba de monitorización de heparina POC proporciona resultados rápidos
- Nueva herramienta utiliza aprendizaje profundo para terapia de precisión contra cáncer
- Prueba diagnóstica complementaria identifica cáncer de mama y de vías biliares con HER2 ultrabajo
- Análisis sanguíneo predice eficacia de inmunoterapia en cáncer mamario triple negativo
- Pruebas genéticas simples podrían predecir éxito del tratamiento de esclerosis múltiple
- Nueva firma genética predice respuesta a inmunoterapia en cánceres renales avanzados
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
- Nuevo método diagnóstico confirma sepsis de forma más temprana
- Nuevos marcadores podrían predecir riesgo de infección grave por clamidia
- Espectroscopia portátil detecta de forma rápida y no invasiva bacterias en fluido vaginal
- Prueba de saliva basada en CRISPR detecta tuberculosis en esputo
- Muestras de cápsulas inspiradas en corales ocultan bacterias intestinales
- Tecnología de diagnóstico rápido detecta infecciones del tracto respiratorio inferior en muestras de aliento
- Sensor de grafeno utiliza muestra de aliento para identificar diabetes y prediabetes en minutos
- Parche de sudor inalámbrico podría usarse como prueba diagnóstica para fibrosis quística
- Nuevo método mejora fiabilidad de la IA con aplicaciones en diagnóstico médico
- VedaBio se asocia con Mammoth Biosciences para expandir diagnósticos basados en CRISPR
- Werfen y VolitionRx se asocian para adelantar pruebas diagnósticas del síndrome antifosfolípido
- Siemens Healthineers y Carna Health se asocian para impulsar innovación en cuidado renal
- Tempus AI adquiere empresa de patología digital Paige
- Vircell lanza nuevo sitio web para sus productos de control molecular
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Adelanto en VSG amplía estabilidad de muestras sanguíneas de 4 a 28 horas
- Análisis patológico preciso mejora resultados del tratamiento del fibrosarcoma en adultos
- Estudio clinicopatológico respalda exclusión del carcinoma seroso cervical de clasificación de OMS
- Sistema con IA compatible con dispositivos móviles revolucionará diagnóstico de malaria
- Microscopio compacto con IA permite puntuación del cáncer rápida y rentable