Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunología
PatologíaTecnologíaIndustria
Eventos
Webinars

- Vesículas extracelulares se relacionan con riesgo de insuficiencia cardíaca en pacientes con ERC
- Estudio compara rendimiento analítico de ensayos cuantitativos de antígeno de superficie de hepatitis B
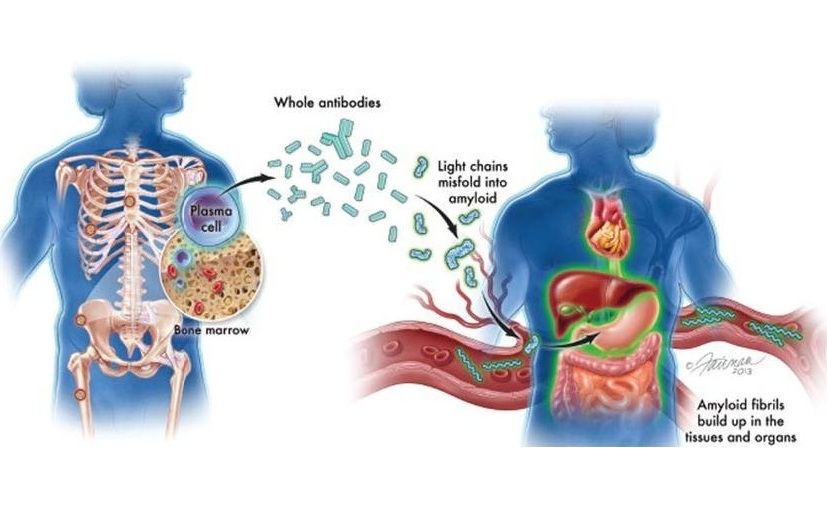
- Análisis sanguíneo podría predecir e identificar recaídas tempranas en pacientes con mieloma
- Sistema compacto de imágenes Raman detecta señales tumorales sutiles
- Monitoreo no invasivo de glucosa reemplaza pinchazos en dedo en diabéticos
- El dispositivo "Lab-On-A-Disc" abre el camino hacia biopsias líquidas más automatizadas
- Análisis de ADN dos en uno mejora precisión diagnóstica y ahorra tiempo y costes
- Nueva herramienta mapea cambios cromosómicos en células cancerosas para predecir evolución de tumores
- Análisis sanguíneo identifica pacientes con cáncer mamario inflamatorio con mayor riesgo de metástasis cerebral
- Biomarcadores de Parkinson recientemente identificados permitirán diagnóstico temprano mediante análisis sanguíneo
- Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
- Prueba rápida y sencilla podría revolucionar transfusiones de sangre
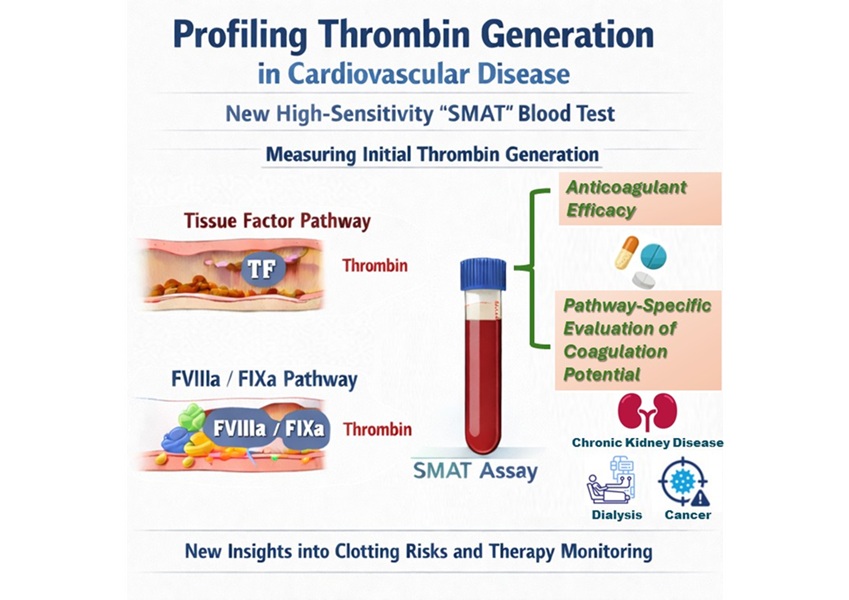
- Análisis de sangre muy sensible mejora evaluación del riesgo de coagulación en pacientes cardíacos
- Algoritmo de IA distingue eficazmente subtipos de alfa talasemia
- Pruebas de MRD podrían predecir supervivencia en pacientes con leucemia
- Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
- Biopsia líquida ultrasensible demuestra eficacia para predecir respuesta a inmunoterapia
- Análisis sanguíneo identifica pacientes con cáncer de colon que se benefician de AINE
- Análisis sanguíneo podría detectar efectos adversos de inmunoterapia
- Análisis de sangre rutinario puede predecir mayor beneficiario de terapia con células T CAR
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
- Tecnología de biosensores de aptámeros transforma detección de virus
- Análisis de sangre de rutina y modelos de IA podrían predecir preeclampsia y anemia
- Sensores generados por IA abren caminos para detección temprana del cáncer
- Prueba sanguínea pionera detecta cáncer de pulmón mediante imágenes infrarrojas
- Nueva colaboración lleva espectrometría de masas automatizada a pruebas de laboratorio de rutina
- Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
- WHX Labs Dubai reunirá expertos mundiales en resistencia a antimicrobianos en cumbre inaugural de líderes de RAM
- BD y Penn Institute colaboran para avanzar en inmunoterapia mediante citometría de flujo
- Abbott adquiere Exact Sciences, empresa de detección de cáncer
- IA mejora detección de mutaciones genéticas en diagnóstico del cáncer
- Biopsia de piel ofrece nuevo método diagnóstico para enfermedades neurodegenerativas
- Método rápido sin etiquetas identifica células cancerosas agresivas
- Nuevo método de rayos X promete avances en histología
- Técnica de perfilado de células individuales orienta detección del cáncer

 Expo
Expo
- Vesículas extracelulares se relacionan con riesgo de insuficiencia cardíaca en pacientes con ERC
- Estudio compara rendimiento analítico de ensayos cuantitativos de antígeno de superficie de hepatitis B
- Análisis sanguíneo podría predecir e identificar recaídas tempranas en pacientes con mieloma
- Sistema compacto de imágenes Raman detecta señales tumorales sutiles
- Monitoreo no invasivo de glucosa reemplaza pinchazos en dedo en diabéticos
- El dispositivo "Lab-On-A-Disc" abre el camino hacia biopsias líquidas más automatizadas
- Análisis de ADN dos en uno mejora precisión diagnóstica y ahorra tiempo y costes
- Nueva herramienta mapea cambios cromosómicos en células cancerosas para predecir evolución de tumores
- Análisis sanguíneo identifica pacientes con cáncer mamario inflamatorio con mayor riesgo de metástasis cerebral
- Biomarcadores de Parkinson recientemente identificados permitirán diagnóstico temprano mediante análisis sanguíneo
- Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
- Prueba rápida y sencilla podría revolucionar transfusiones de sangre
- Análisis de sangre muy sensible mejora evaluación del riesgo de coagulación en pacientes cardíacos
- Algoritmo de IA distingue eficazmente subtipos de alfa talasemia
- Pruebas de MRD podrían predecir supervivencia en pacientes con leucemia
- Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
- Biopsia líquida ultrasensible demuestra eficacia para predecir respuesta a inmunoterapia
- Análisis sanguíneo identifica pacientes con cáncer de colon que se benefician de AINE
- Análisis sanguíneo podría detectar efectos adversos de inmunoterapia
- Análisis de sangre rutinario puede predecir mayor beneficiario de terapia con células T CAR
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
- Tecnología de biosensores de aptámeros transforma detección de virus
- Análisis de sangre de rutina y modelos de IA podrían predecir preeclampsia y anemia
- Sensores generados por IA abren caminos para detección temprana del cáncer
- Prueba sanguínea pionera detecta cáncer de pulmón mediante imágenes infrarrojas
- Nueva colaboración lleva espectrometría de masas automatizada a pruebas de laboratorio de rutina
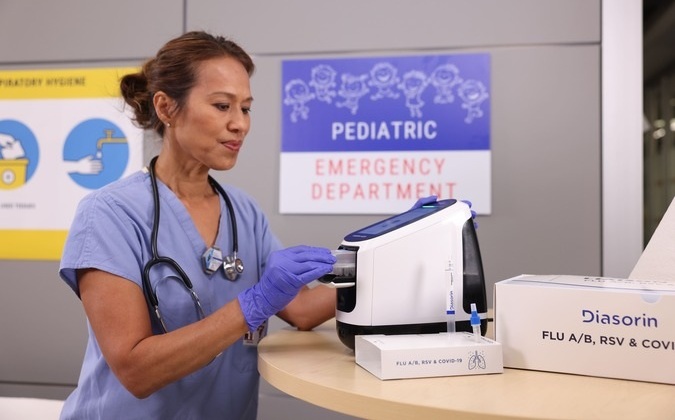
- Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
- WHX Labs Dubai reunirá expertos mundiales en resistencia a antimicrobianos en cumbre inaugural de líderes de RAM
- BD y Penn Institute colaboran para avanzar en inmunoterapia mediante citometría de flujo
- Abbott adquiere Exact Sciences, empresa de detección de cáncer
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- IA mejora detección de mutaciones genéticas en diagnóstico del cáncer
- Biopsia de piel ofrece nuevo método diagnóstico para enfermedades neurodegenerativas
- Método rápido sin etiquetas identifica células cancerosas agresivas
- Nuevo método de rayos X promete avances en histología
- Técnica de perfilado de células individuales orienta detección del cáncer








.jpg)