Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Webinars

- Nariz electrónica detecta signos tempranos de cáncer de ovario en sangre
- Simple análisis sanguíneo ofrece nueva vía para evaluación del Alzheimer en atención primaria
- Analizadores hospitalarios existentes pueden identificar productos médicos líquidos falsos
- Método rápido de análisis sanguíneo permite decisiones más seguras en emergencias por medicamentos
- Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
- Nueva prueba detecta Alzheimer al analizar formas alteradas de proteínas en sangre
- Nuevos marcadores diagnósticos para esclerosis múltiple descubiertos en líquido cefalorraquídeo
- ADN libre circulante predice infecciones del torrente sanguíneo en niños con leucemia
- Estudio utiliza muestras de sangre para identificar enfermedades años antes de que aparezcan
- Método basado en microARN predice la enfermedad renal crónica y riesgo cardiovascular
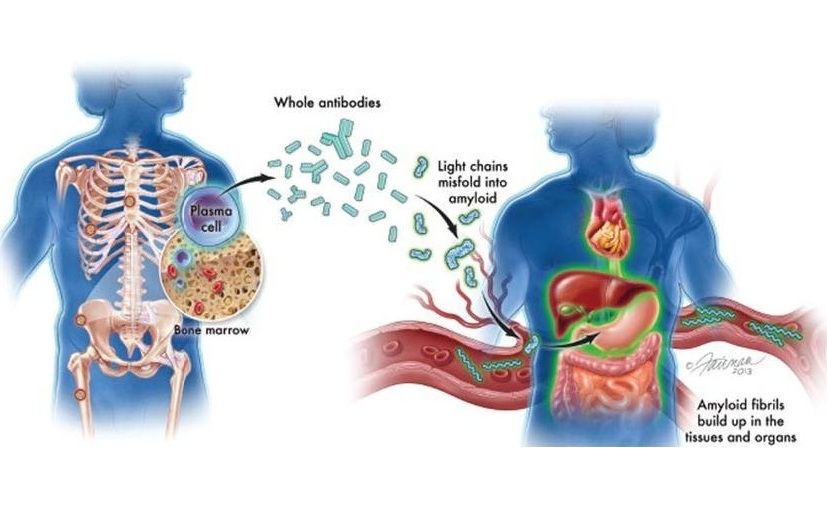
- Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
- Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
- Prueba rápida y sencilla podría revolucionar transfusiones de sangre
- Análisis de sangre muy sensible mejora evaluación del riesgo de coagulación en pacientes cardíacos
- Algoritmo de IA distingue eficazmente subtipos de alfa talasemia
- Nuevo biomarcador predice respuesta a quimioterapia en cáncer de mama triple negativo
- Análisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
- Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
- Biopsia líquida ultrasensible demuestra eficacia para predecir respuesta a inmunoterapia
- Análisis sanguíneo identifica pacientes con cáncer de colon que se benefician de AINE
- Virus intestinales ocultos vinculados al riesgo de cáncer colorrectal
- Panel de tres pruebas para detección de infecciones por duelas del hígado
- Prueba rápida promete respuestas inmediatas a infecciones resistentes a medicamentos
- Tecnología con CRISPR neutraliza bacterias resistentes a antibióticos
- Nuevo estudio identifica características del microbioma intestinal asociadas con enfermedad de Alzheimer
- “Relojes” de análisis sanguíneos predicen inicio de síntomas del Alzheimer
- Biomarcador de IA predice riesgo de cáncer de hígado
- Tecnología robótica para extracciones de sangre diagnósticas automatizadas
- ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
- Tecnología de biosensores de aptámeros transforma detección de virus
- QuidelOrtho colabora con Lifotronic para ampliar cartera global de inmunoensayos
- WHX Labs en Dubái resalta habilidades de liderazgo que forman laboratorios de próxima generación
- Prueba de cáncer cervical con IA lista para lanzamiento en América Latina
- Nueva colaboración lleva espectrometría de masas automatizada a pruebas de laboratorio de rutina
- Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
- Sistema de recolección de orina mejora precisión y eficiencia del diagnóstico
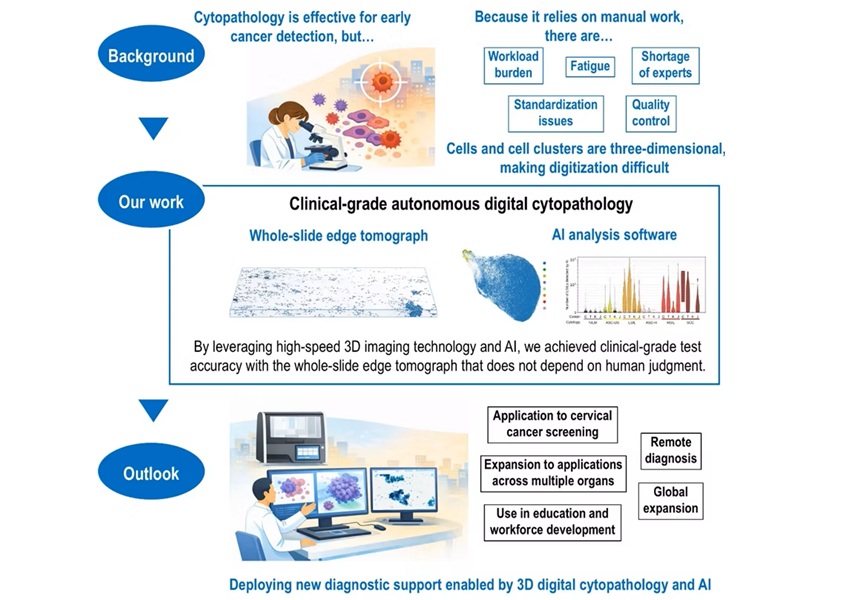
- Escaneo 3D con IA acelera detección del cáncer
- Clasificador de muestras únicas predice subtipos de fibroblastos asociados al cáncer
- Plataforma de IA estandariza flujo de trabajo de microscopía de frotis de tuberculosis
- Herramienta de IA predice complicaciones del trasplante mediante biomarcadores sanguíneos antes de síntomas

 Expo
Expo
- Nariz electrónica detecta signos tempranos de cáncer de ovario en sangre
- Simple análisis sanguíneo ofrece nueva vía para evaluación del Alzheimer en atención primaria
- Analizadores hospitalarios existentes pueden identificar productos médicos líquidos falsos
- Método rápido de análisis sanguíneo permite decisiones más seguras en emergencias por medicamentos
- Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
- Nueva prueba detecta Alzheimer al analizar formas alteradas de proteínas en sangre
- Nuevos marcadores diagnósticos para esclerosis múltiple descubiertos en líquido cefalorraquídeo
- ADN libre circulante predice infecciones del torrente sanguíneo en niños con leucemia
- Estudio utiliza muestras de sangre para identificar enfermedades años antes de que aparezcan
- Método basado en microARN predice la enfermedad renal crónica y riesgo cardiovascular
- Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
- Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
- Prueba rápida y sencilla podría revolucionar transfusiones de sangre
- Análisis de sangre muy sensible mejora evaluación del riesgo de coagulación en pacientes cardíacos
- Algoritmo de IA distingue eficazmente subtipos de alfa talasemia
- Nuevo biomarcador predice respuesta a quimioterapia en cáncer de mama triple negativo
- Análisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
- Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
- Biopsia líquida ultrasensible demuestra eficacia para predecir respuesta a inmunoterapia
- Análisis sanguíneo identifica pacientes con cáncer de colon que se benefician de AINE
- Virus intestinales ocultos vinculados al riesgo de cáncer colorrectal
- Panel de tres pruebas para detección de infecciones por duelas del hígado
- Prueba rápida promete respuestas inmediatas a infecciones resistentes a medicamentos
- Tecnología con CRISPR neutraliza bacterias resistentes a antibióticos
- Nuevo estudio identifica características del microbioma intestinal asociadas con enfermedad de Alzheimer
- “Relojes” de análisis sanguíneos predicen inicio de síntomas del Alzheimer
- Biomarcador de IA predice riesgo de cáncer de hígado
- Tecnología robótica para extracciones de sangre diagnósticas automatizadas
- ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
- Tecnología de biosensores de aptámeros transforma detección de virus
- QuidelOrtho colabora con Lifotronic para ampliar cartera global de inmunoensayos
- WHX Labs en Dubái resalta habilidades de liderazgo que forman laboratorios de próxima generación
- Prueba de cáncer cervical con IA lista para lanzamiento en América Latina
- Nueva colaboración lleva espectrometría de masas automatizada a pruebas de laboratorio de rutina
- Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Sistema de recolección de orina mejora precisión y eficiencia del diagnóstico
- Escaneo 3D con IA acelera detección del cáncer
- Clasificador de muestras únicas predice subtipos de fibroblastos asociados al cáncer
- Plataforma de IA estandariza flujo de trabajo de microscopía de frotis de tuberculosis
- Herramienta de IA predice complicaciones del trasplante mediante biomarcadores sanguíneos antes de síntomas














 (3) (1).png)